In den letzten Jahren hat sich im medizinischen Forschungsbereich der Trend etabliert, populations-basierte Studien mit einer sehr großen Anzahl an Versuchspersonen durchzuführen. Je mehr Probanden untersucht werden, desto mehr Daten stehen den Forschern für ihre Analysen zur Verfügung. Das ist die Voraussetzung um Fragen wie „welchen Einfluss hat der Lebensstil auf die Gesundheit“ oder „welchen Zusammenhang gibt es zwischen der Genetik und bestimmten Erkrankungen“ erforschen zu können. Deshalb gibt es weltweit viele Konsortien, die darauf abzielen, möglichst große und repräsentative Stichproben zu erheben, um besonders gut gesicherte Rückschlüsse und Zusammenhänge im Bereich der Gesundheitsforschung ziehen zu können. Den größten Gesundheitsdatensatz stellt die britische UK Biobank Kohorte dar, die in den vergangenen Jahren gesundheitsbezogene Daten von 500.000 Probanden im Alter von 40 bis 69 Jahren erhoben hat, um neue wissenschaftliche Erkenntnisse über häufige und lebensbedrohliche Krankheiten – wie Krebs, Herzerkrankungen und Schlaganfall – zu gewinnen und so die Gesundheit der Bevölkerung zu verbessern. Zusätzlich werden seit 2014 von prospektiv 100.000 Probanden dieser Stichprobe unter anderem Magnetresonanztomographieaufnahmen des Gehirns, des Herzens und des Abdomens erstellt. Solche umfassenden Datensätze, die Forschern weltweit zugänglich gemacht werden, ermöglichen es, die Auswirkungen verschiedener Einflüsse auf die Entwicklung unterschiedlichste Erkrankungen genauer zu beleuchten.
Obwohl dieser Trend sehr förderlich ist, stellen die Speicherung und Verarbeitung dieser enormen Datenmengen die Forscher auch vor komplexe Probleme. Die erste Schwierigkeit betrifft die Beschaffung der Daten. Das Herunterladen der Daten aus einer Cloud würde Wochen oder sogar Monate dauern, weshalb sie häufig auf großen Festplatten gespeichert und per Kurier verschickt werden.
Wenn die Forscher die Daten endlich erhalten haben, besteht die nächste Herausforderung darin, einen Computer zu finden, der den Anforderungen der Speicherung gerecht wird. Sogar hochrangige Supercomputer könnten entweder mit dem benötigten Speicherplatz oder der Anzahl der abzuspeichernden Dateien überfordert sein. Wenn ein Datensatz die rechnergestützten Kapazitätsgrenzen in unterschiedlichen Dimensionen überschreitet, erschwert dies die Prozessierung ungemein.
Eine weitere Anforderung an die Wissenschaft ist, dass Forschungsergebnisse besonders reproduzierbar, also wiederholbar und vertrauenswürdig sein sollen. Erst wenn eine Replikationsstudie die Berechnungen einer anderen Studie wiederholt und zu ähnlichen bzw. gleichen Ergebnissen kommt wie die Erst-Studie, erlangt diese Glaubwürdigkeit. Das ist allerdings bei großen Datensätzen besonders schwierig, da sie oft besonders strengen Datenschutzbestimmungen unterliegen und sie dadurch nicht ohne Weiteres mit anderen Forschern geteilt werden dürfen. Zudem sind viele der gängigen Softwaretools nicht öffentlich zugänglich, weshalb sie nicht jedem Wissenschaftler zur Verfügung stehen, was wiederum die Replikation von Studien erschwert.
Zusammenfassend werden Wissenschaftler bei der Analyse umfangreicher Datensätze mit einigen Herausforderungen konfrontiert. Die Beschaffung der Daten ist bereits relativ aufwendig. Zudem können die Anforderungen des Datensatzes die Hardware überfordern, weshalb ein Computer gefunden werden muss, der leistungsfähig genug ist und diesem Bedarf gerecht wird. Die nächste Schwierigkeit besteht darin, die Ergebnisse überprüfbar und transparent darzulegen, sodass andere Forscher die durchgeführten Analyseschritte nachvollziehen und sie wiederholen können, um die Ergebnisse zu verifizieren.
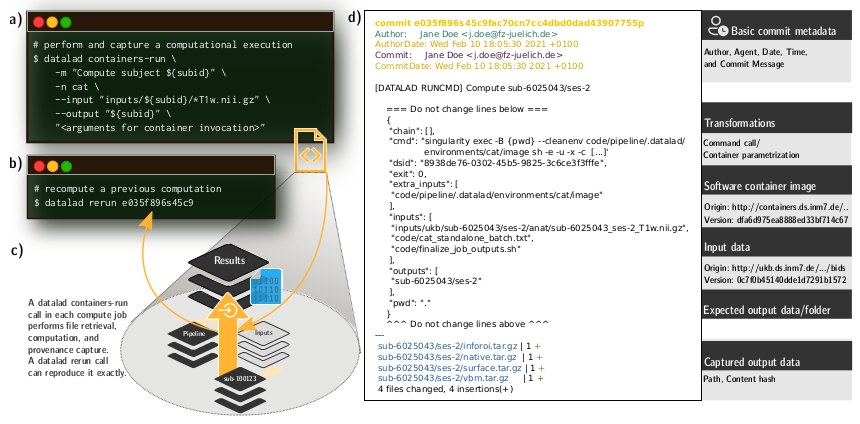
Um dafür eine Lösung zu finden, haben Kollegen unseres Instituts basierend auf dem Neurobildgebungsdatensatz der UK Biobank, der zum Zeitpunkt des Projekts die Daten von etwa 42.000 Probanden umfasste, eine ganz besondere Prozessierungstechnik entwickelt. Für diese Technik haben sie ein System verwendet, das die gesamte Provenienz, also die Dokumentation darüber, woher das Datenmaterial stammt und mit welchen Prozessen und Methoden (Code, Softwaretools etc.) es verarbeitet wurde, erfasst. Eine solch detaillierte Dokumentation ist wichtig, um Reproduzierbarkeit zu ermöglichen. Die Besonderheit hier besteht darin, dass die Provenienz vom Computer ausgelesen und die vergangene Analyse automatisch wiederausgeführt werden kann, ohne mit den beteiligten Wissenschaftlern Rücksprache über die verwendeten Verarbeitungsschritte und deren Reihenfolge halten zu müssen. Das befähigt uns beispielsweise dazu, Analysen nach einer Erweiterung des zugrunde liegenden Datensatzes automatisch zu wiederholen oder die Reproduzierbarkeit unserer Ergebnisse zu prüfen.
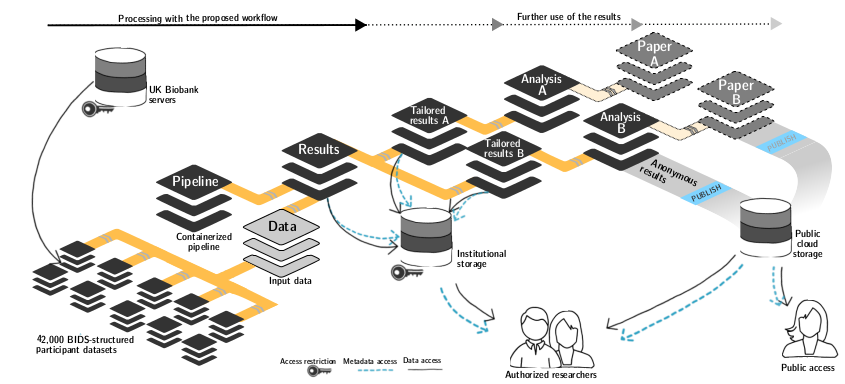
Für die Entwicklung dieses Workflows haben unsere Wissenschaftler auf etablierte Softwarewerkzeuge aus Industrie und Wirtschaft (bspw. Git, HTCondor, SLURM) sowie lokal am Forschungszentrum Jülich entwickelte Software zum Datenmanagement (DataLad) zurückgegriffen. Dadurch werden Erkenntnisse aus der Industrie mit Erkenntnissen aus der Wissenschaft vereint, um unsere Forschung besonders reproduzierbar und skalierbar zu machen und dadurch insgesamt ihre Qualität zu steigern.
Als Grundlage für die Prozessierungstechnik haben unsere Wissenschaftler Software Container gewählt. Dies sind Softwareumgebungen, die ein minimales Betriebssystem und alle wichtigen Programme, die für eine Analyse benötigt werden, enthalten. In einem solchen Software Container können Analysen durchgeführt werden, ohne dass die notwendigen Programme auf dem Computer installieren sein müssen. Außerdem können sie mit anderen Personen geteilt werden, um ihnen die richtige Softwareumgebung einfach zur Verfügung zu stellen. Zum einen können dadurch für die Analyse auch nicht freiverfügbare Softwaretools genutzt werden und zum anderen sind die Analysen Hardware unabhängig, sodass Replikationsstudien nicht nur auf großen Supercomputern, sondern auch auf Laptops durchgeführt werden können.
Der gesamte Prozess ist komplett generisch, also auf alle möglichen Forschungsbereiche (wie z. B. Geowissenschaften, Politikwissenschaften, Wirtschaftswissenschaften etc.) anwendbar und nicht an bestimmte Datensätze, Analyseschritte, oder Softwaretools gebunden. Die Anwendung an einem so großen Datensatz, wie dem der UK Biobank, zeigt, dass das erstellte und öffentlich zur Verfügung gestellte Tool sehr gut auf wirklich große Datensätze anwendbar ist.
Zu Visualisierungszwecken haben unsere Kollegen ein Video erstellt, das eindrucksvoll die Dimension einer Datenanalyse dieses Ausmaßes und auch die immense Leistungsfähigkeit der rechnergestützten Infrastruktur, die am Forschungszentrum Jülich existiert, demonstriert: Zwischen Minute 00:20 und 01:20 ist die Analyse auf dem high-throughput compute cluster unseres Instituts visualisiert, und ab Minute 01:20 ist die Prozessierung auf dem Supercomputer JURECA dargestellt. Hier kommen Sie zum Video: https://www.youtube.com/watch?v=UsW6xN2f2jc
Um anderen Neurowissenschaftlern die Komplexität der Datenanalysen, die an unserem Institut durchgeführt werden, näher zu bringen, haben wir das Video bei der diesjährigen Brain Art Competition des alljährlichen Meetings der Organization for Human Brain Mapping (OHBM) eingereicht und in der Kategorie Video/Animation den zweiten Platz belegt!
Das Preprint zur Publikation ist hier zu finden: https://www.biorxiv.org/content/10.1101/2021.10.12.464122v1

No Comments
Be the first to start a conversation