Eine bioökonomische Nutzung von Pflanzen und anderen Organismen zur Produktion von nachhaltigen Ressourcen wird durch die genetischen Grundlagen der Organismen beeinflusst. Die Erzeugung von Daten über Genome und auch von Daten zu der aktiven Ausprägung des Genoms (seiner „Expression“) ist deshalb wichtig, um die dahinterliegenden Prozesse zu verstehen und zu verbessern oder an den Bedarf der bioökonomischen Nutzung anzupassen wie z. B. in der Züchtung oder Auswahl von geeigneten Nutzpflanzen. Komplexe Daten, die bei Untersuchungen von Genomen und seiner Ausprägung entstehen, müssen in Datenbanken und durch Visualisierungen verwaltet, verknüpft und interpretierbar gemacht werden. Das alles leistet die Bioinformatik mit verschiedenen Techniken, Algorithmen und Programmen, am IBG-4 zum Beispiel in der Forschung zur Nutzung von Pflanzen zur Gewinnung speziellen industriell interessanten Pflanzeninhaltsstoffen in dem Projekt TaReCa.
Quelle: Kathryn Dumschott, Rainer Knauf, Björn Usadel
Am IBG-4 nutzen wir die Bioinformatik und das Wissen um die genetische Variabilität und die dynamische Anpassungsfähigkeit von Pflanzen für die gezielte Anwendung in der Bioökonomie. Wir untersuchen gemeinsam mit unseren Partnern, wie nutzbringende Inhaltsstoffe in Pflanzen gesteigert werden können (TaReCa), wie Nutzpflanzen robuster oder effektiver gemacht werden können (TEOSINTE, CassavaStore, BreedPatH), und wie bestimmte Gene z. B. aus Algen biotechnologisch genutzt werden können (HaloENZ). Wir werden in Zukunft aktuell aus unserer Forschung und aus Kooperations-Projekten und den Verbünden zu Bioinformatik, Datenbanken, und Plattformen zur Darstellung von Daten (de.NBI Deutsches Netzwerk für Bioinformatik-Infrastruktur, EPPN2020, EMPHASIS, EOSC-Life, NFDI) berichten und dabei einzelne Begriffe und Methoden der Bioinformatik erläutern.
Nutzung von Pflanzeninhaltsstoffen für die Bioökonomie
Pflanzen produzieren viele nützliche Inhaltsstoffe, angefangen damit, dass sie als Kohlenhydrat- Fett- und Eiweißquelle für unsere Ernährung und zur energetischen Nutzung als Kraftstoff oder Brennstoffe dienen und wichtige Vitamine zur Ernährung beitragen. Weiterhin können auch sogenannte Sekundär- oder Spezialmetabolite aus Pflanzen große Bedeutung haben. Heilpflanzen und Gewürzpflanzen, wie beispielsweise Kamille, Basilikum oder Arnica sind allgemein bekannt, und werden wegen ihren speziellen Inhaltsstoffen kommerziell angebaut. Auch wirtschaftlich sind solche Pflanzeninhaltsstoffe wichtig, z. B. in der Kosmetikindustrie, der Pharmaindustrie, und als Nahrungsergänzungsmittel. Das IBG-4 ist an Projekten beteiligt, in denen daran gearbeitet wird, die Mengen und die Extrahierbarkeit von Inhaltsstoffe aus Pflanzen zu verbessern und zu nutzen, wie im Projekt TaReCa:
TaReCa – Nutzung von Restpflanzen aus dem Gewächshaus zur Gewinnung von Flavonoiden
„Tailoring of secondary metabolism in horticultural residuals and cascade utilization for a resource efficient production of valuable bioactive compounds”– Im Projekt TaReCa untersuchen wir Inhaltsstoffe der Paprikapflanze. Paprikapflanzen werden in Gewächshäusern zur Produktion der Paprikafrüchte mehrere Monate lang kultiviert, im Herbst werden die letzten Früchte abgeerntet und die verbleibenden großen Pflanzen werden kompostiert. Die Projektidee des Projektes TaReCa ist, diese verbleibenden Pflanzen noch weiter zu nutzen. Dabei sollen die Blätter der Pflanzen zur Extraktion von nutzbaren speziellen („sekundären“) Pflanzeninhaltsstoffen (z.B. Flavonoide, Cynarosid und Graveobiosid A) verwendet werden. Pflanzeninhaltstoffe wie die in diesem Projekt untersuchten Flavonoide sind oft anti-oxidativ und haben vielfach beschriebene Funktionen, die sowohl pharmazeutisch als auch anders industriell nutzbar sind. Pflanzen produzieren diese Spezialmetabolite häufig als chemischen Schutz vor Umweltstressen, deshalb untersucht das Team von TaReCa die Möglichkeit, die Paprikapflanzen durch gezieltes Anlegen von Stressen zur erhöhten Produktion von Inhaltsstoffen anzuregen, die dann in höheren Konzentrationen aus den Pflanzenrestmassen extrahiert werden können. Die Veränderung der Konzentration von Flavonoiden und der Zielmetabolite Cynarosid und Graveobiosid A durch die Stressbehandlungen werden gemessen, Genexpression wird bei erhöhten Konzentrationen der untersuchten Inhaltsstoffe mit dem Ziel der Identifizierung der Biosyntheseweg-Gene und deren Regulationsmechanismen analysiert. TaReCa ist ein interdisziplinäres Verbundprojekt mit Partnern an IBG-2 und IBG-4 und an der Uni-Bonn und der RWTH Aachen.
Das Projekt wird noch bis Oktober 2020 vom BMBF im Rahmen der Förderung „Maßgeschneiderte biobasierte Inhaltsstoffe für eine wettbewerbsfähige Bioökonomie“ im Rahmen der „Nationalen Forschungsstrategie BioÖkonomie 2030“ gefördert.

Paprika im Gewächshaus. Quelle: Anika Wiese-Klinkenberg
| Box 1) Was sind Gene und was bedeutet Gen-Expression
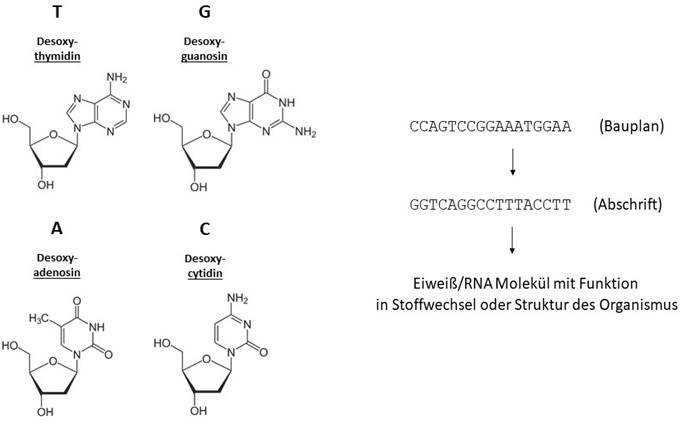
Die genetischen Programme aller Organismen bestehen aus Genen , die aus den 4 verschiedenen DNA-Bausteinen zusammengesetzt, Informationen („Baupläne“) zur Erzeugung von Proteinen oder RNA Molekülen enthalten, die am Ende alle Prozesse wie Wachstum, Stoffwechsel, Fortpflanzung, Stressreaktionen und die Produktion von Inhaltsstoffen umsetzen. Höhere Organismen haben oft mehrere zehntausend Gene, die Baupläne für Funktionen in grundliegenden oder speziellen Prozessen enthalten. Oft sind mehrere Gene an einzelnen Teilen von Strukturen oder biochemischen Stoffwechselwegen beteiligt. Diese Gene zu kennen und ihre Funktion vorherzusagen ist ein wichtiger Bestandteil in der bioökonomischen Nutzung von Organismen. Gene für alle Prozesse sind immer und in allen Zellen vorhanden und werden aber nur in z. B. bestimmten Geweben (wie z. B. in der Pflanzenwurzel) oder zu bestimmten Gegebenheiten wie z. B. Photosynthesegene am Tag, oder Gene zum Schutz gegen Stress bei bestimmten Stressen „exprimiert“ oder „angeschaltet“. Das bedeutet die DNA-Baupläne werden dann tatsächlich umgesetzt und zeigen eine Ausprägung im Organismus, wenn und wo sie benötigt werden. Diese Genexpression und auch die Prozesse, die sie steuern zu kennen und zu verstehen ist wichtig, um Organismen bioökonomisch zu nutzen. |
| Box 2) Bioinformatik
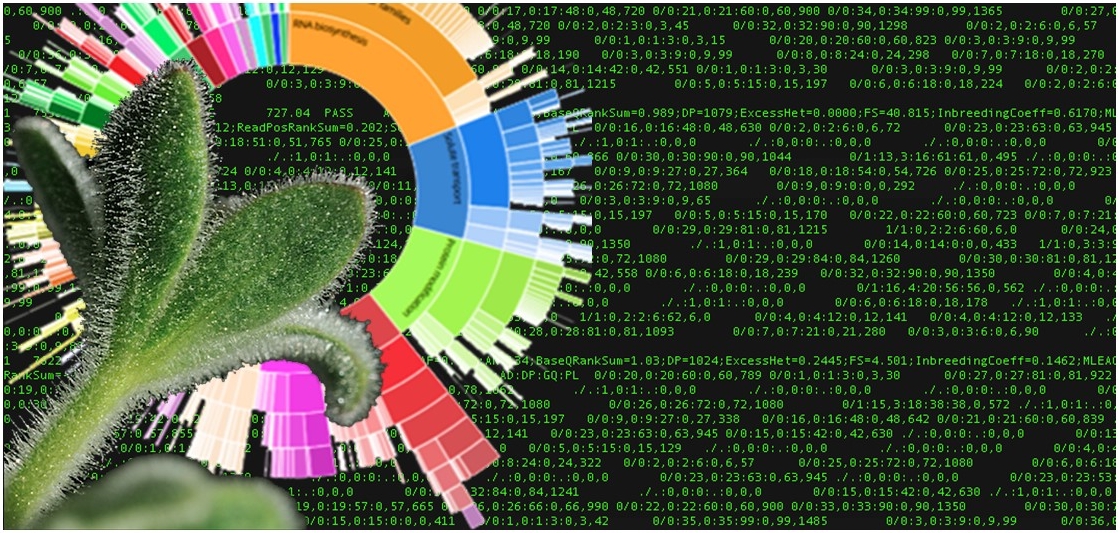
Das Vorhandensein von bis zu mehreren 10.000 Genen in den verschiedenen Organismen und die Techniken mit denen diese Gene „gelesen“ (sequenziert) werden, haben zur Folge, dass die Erforschung der Gene und deren Umsetzung in RNA und weiter in Proteine und der teilweise daraus gesteuerte Stoffwechsel (Metabolite) mit vielen komplexen Daten einhergeht. Deshalb sind computer-gestützte Analysen und Prozesse unabdingbar, um die Gene und ihre Gesamtheit (Genome) zu verstehen. Datenbanken werden benötigt, um die Informationen zu den Genen zugänglich und begreifbar zu machen. Es braucht Forschung mit computer-gestützte Analysen, um den einzelnen Genen ihre bekannte oder mögliche Funktion zuzuordnen. Die Computerprogrammierungen, die für die Analyse solcher und weiterer biologischen Prozesse erstellt werden und die Wissenschaft, die mit diesen computer-gestützten Analysen durchgeführt wird, nennt man Bioinformatik. |
| Box 3) Lösungen zur Analyse und Visualisierungen von Daten
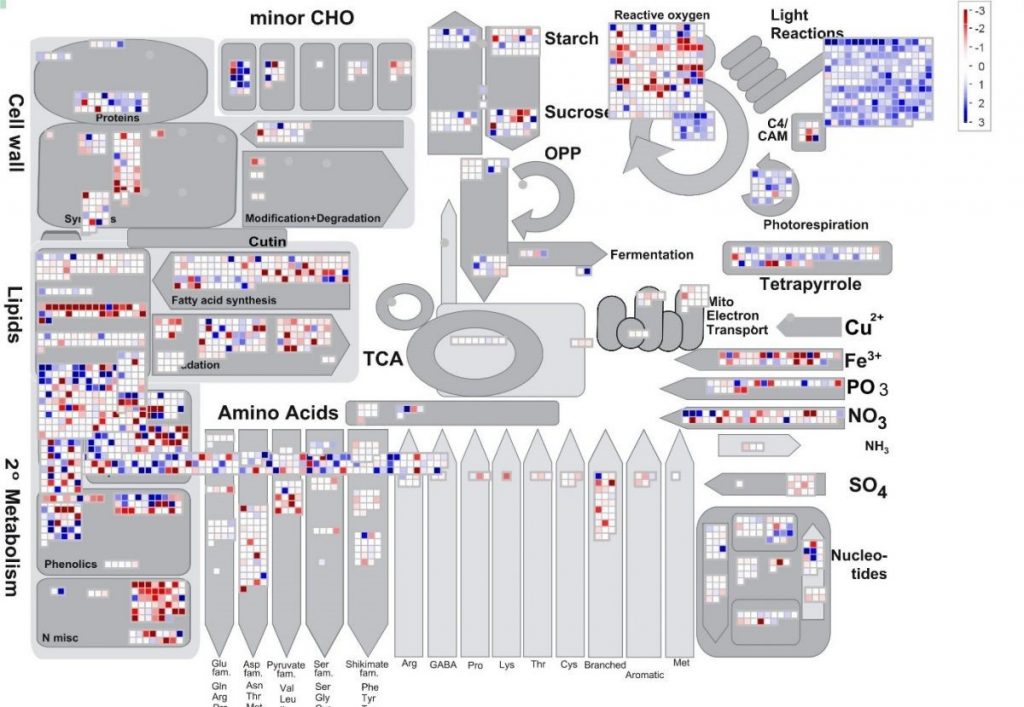
Wegen der vielen und komplexen Daten, die oft nach den unterschiedlichen angewendeten bioinformatischen Analysen entstehen, ist es nötig diese nachvollziehbar zusammenzufassen und darzustellen. Hierzu werden Computerprogramme entwickelt, die helfen solche Daten zu validieren, zu analysieren und gebündelt darzustellen. So kann zum Beispiel die Expression von Genen auf verschieden Arten ausgewertet und Prozessen zugeordnet dargestellt werden. So kann der Wissenschaftler am Ende das Zusammenspiel der Genexpression und dessen Regulation verstehen und analysieren. |

No Comments
Be the first to start a conversation