Marine Naturstoffe weisen im Vergleich zu Naturstoffen aus terrestrischen Systemen oft eine größere Anzahl an halogenierten Verbindungen auf. Halogene sind u.a. die Elemente Fluor, Chlor, Brom und Iod. Diese werden oft an pharmazeutische Moleküle angehängt, um deren Bio-Aktivität und Wirkung zu verbessern. Dabei ist es wichtig, dass die Halogene gezielt an die richtige Stelle der Moleküle gekoppelt werden. Das kann man sehr gut mit Enzymen sicherstellen, die Moleküle und Halogene kontrolliert binden und zusammenführen. Deshalb untersucht das BioSC-Seed-Fund Projekt HaloEnz unter anderem den Lappentang (Palmaria palmata), den Knotentang (Ascophyllum nodosum) und auch den Meersalat (Ulva lactuca/Ulva australis) um Enzyme zu finden, die die Halogenierung von Molekülen ermöglichen. Auch einzellige Algen wurden in der Arbeitsgruppe von Prof. Usadel sequenziert, um Enzyme zu untersuchen.
„Haloenz“, ein Projekt mit Förderung aus dem Bioeconomy Science Center (BioSC)
Das vom Bioeconomy Science Center (BioSC) geförderte Projekt HaloEnz ist im September letzten Jahres gestartet und wird von Seiten des IBG-4 durch Frau Dr. Kathryn Dumschott bearbeitet. Aufgabe des IBG-4 ist die Suche nach Genen, die für sogenannte „Haloenzyme“ kodieren. Hierzu kollaboriert das IBG-4 mit der National University of Ireland Galway wo Makroaalgen auch aus natürlichen Habitaten gesammelt werden. Verschiedene Makroalgen wurden zur Untersuchung ausgewählt, die zunächst im Labor angezogen werden mussten (s. Bild). Jetzt wurde die erste genomische DNA aus verschiedenen Algen extrahiert, um die Genomanalyse, das „Sequenzieren“ durchzuführen. Beim Sequenzieren werden alle Gene eines Organismus abgelesen und dann mit verschiedenen Algorithmen überprüft, in die richtige Reihenfolge sortiert und bestimmten Funktionen zugeordnet. So können die Forscher bestimmte Enzymfunktionen den Gensequenzen oder den daraus „übersetzten“ Aminosäureabfolgen zuordnen. Dabei werden die Baupläne für neue Enzyme in Genomen durch den Vergleich mit bekannten Genen abgeglichen. Einige Haloenzyme und ihre Gene sind bereits bekannt und anhand von Ähnlichkeiten werden zunächst in Datenbanken und dann in den neuen Genomen der Algen weitere Haloenzyme gefunden, die andere spezifische Halogen-Verbindungen durchführen könnten und von den Partnern der Heinrich Heine Universität weiter modelliert und schließlich getestet werden.

Anzucht von Makroalgen, Quelle: K. Dumschott
Das Projekt HaloEnz wird koordiniert von Prof. Dr. Holger Gohlke (Computational Pharmaceutical Chemistry and Molecular Bioinformatics), Heinrich-Heine-Universität Düsseldorf. Weiterer Partner sind Prof. Dr. Jörg Pietruszka und Dr. Thomas Classen, Bioorganic Chemistry, Heinrich-Heine-Universität Düsseldorf.
Auch einzellige Algen wurden in der Arbeitsgruppe von Prof. Usadel sequenziert, um Enzyme zu untersuchen, das Genom von Chlorella sorokiniana und Chlorella vulgaris wurde abgelesen und mit weiteren einzelligen Grünalgen verglichen. C. sorokiniana und C. vulgaris zeigen Unterschiede in der Herstellung von bestimmten Fetten (Glycerolipiden). Die Anwesenheit der Gene für Enzyme die zur Synthese dieser Fettbausteine benötigt werden, wurde deshalb in beiden Algen durch Sequenzierung ihrer Genome überprüft.
Die Daten wurden in der folgenden Veröffentlichung gezeigt: Janka Widzgowski, Alexander Vogel, Lena Altrogge, Julia Pfaff, Heiko Schoof, Björn Usadel, Ladislav Nedbal, Ulrich Schurr, Christian Pfaff; High light induces species specific changes in the membrane lipid composition of Chlorella. Biochem J 17 July 2020; 477 (13): 2543–2559. doi: https://doi.org/10.1042/BCJ20200160
| Box 1: Was sind Enzyme?
Enzyme sind Protein-Katalysatoren, also eigentlich Aminosäureketten, die in besonderer Form vorliegen, in der sie wie passende Schraubenschlüssel Metabolite und Moleküle kontrolliert fassen und dann gezielt verbinden oder umwandeln können. Dabei sind Enzyme oft genauer und auch effektiver in den von ihnen katalysierten Umwandlungen und Verknüpfungen als einfache chemische Prozesse. Die Genauigkeit der Umwandlung ist von großer Bedeutung besonders bei medizinischen Anwendungen. 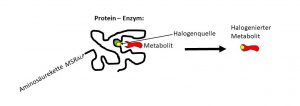 Enzym. Quelle A. Wiese-Klinkenberg
|
| Box 2: Wie wird ein Gen zum Protein?
Es gibt 21 verschiedene „proteinogene“ Aminosäuren, aus denen ein Protein hergestellt werden kann. Diese Aminosäuren werden oft einfach mit einem Buchstaben abgekürzt, hier im Beispiel steht M für Methionin, S für Serin, P für Prolin, R für Arginin und L für Leucin. Dabei stehen die Basen der Gene (A, G, C, T) als sogenanntes Triplett für eine Aminosäure, die hintereinander dann eine Proteinsequenz ergeben (s. Abbildung). Hier bedeutet z. B. die Folge A-T-G, dass ein Methionin eingebaut wird. Alle Aminosäuren haben verschiedene chemische Eigenschaften, die in der Aminosäureabfolge dann sowohl die Form und Stabilität der Proteine beeinflussen als auch deren Aktivitäten als Enzyme.  Translation – vom Gen zum Protein. Quelle: A. Wiese-Klinkenberg
|
| Box 3: Wie findet man „ähnliche“ Gene?
Um gleiche enzymatische Aufgaben zu erfüllen, müssen Enzyme an bestimmten Stellen die gleichen oder ähnlichen Aminosäuren in ihrem Aufbau haben. Da die Enzyme alle von entsprechenden Genen programmiert werden, kann man mit einer bekannten Sequenz mithilfe von Computeralgorithmen nach möglichst ähnlichen Genen suchen, die eben an manchen Stellen anders sind aber an einigen Stellen auch gleich. Die Ausprägung der Ähnlichkeit bestimmt dann, ob man auch eine Ähnlichkeit der Funktion annehmen kann.
|


Super geschriebener und informativer Artikel :-). Eine sehr gute Aufstellung. In diesen Blog werde ich mich noch richtig einlesen ?
Es ist ein sehr informativer Blog, den Sie haben. Vielen Dank für das Teilen mit uns.