Der kaum gebremste Konsum fossiler Ressourcen und die damit verbundenen drastischen Klimaveränderungen mit denen wir Menschen schon heute konfrontiert werden, lassen die Forderungen nach umweltfreundlichen und nachhaltigen Produktionsverfahren in Politik und Gesellschaft immer lauter werden. Hierbei stellt die biotechnologische Produktion auf Basis erneuerbarer Ressourcen eine vielversprechende Alternative zu bisherigen Rohöl-basierten, chemischen Produktionsverfahren dar. Mit Hilfe von Enzymen und Mikroorganismen lassen sich Chemikalien, Kraftstoffen oder Lebensmittel- und Futtermittelzusätzen herstellen. Allerdings werden viele dieser Wertstoffe, ohne die unser modernes Leben nicht möglich wäre, von Mikroorganismen nur in geringsten Mengen produziert. Somit können solche mikrobiellen Prozesse nicht mit den etablierten Verfahren auf Basis von fossilen Rohstoffen konkurrieren und werden nicht im industriellen Maßstab eingesetzt.
Um dies zu ändern versuchen Forscher die mikrobielle Biosynthese eines Produktes durch gezielte genetische Veränderung des Erbguts des produzierenden Mikroorganismus zu verbessern und somit wirtschaftlicher zu machen. Da aber der Stoffwechsel selbst einfachster Bakterien noch nicht vollständig verstanden ist, weiß man anfänglich oft nicht wie die DNA verändert werden muss um die Synthese des gewünschten Produktes zu erhöhen. Aus diesem Grund greift man bei der Entwicklung von Produktionsstämmen immer noch auf klassische Techniken wie das Mutagenese/Screening Verfahren zurück. Bei einer Mutagenese induziert man zunächst zufällige Veränderung der Erbinformation durch Zugabe von DNA-verändernden Chemikalien oder dem Einsatz von energiereicher Strahlung (UV- oder radioaktive Strahlung). Unter vielen Millionen der so geschaffenen genetischen unterschiedlichen Varianten eines Ausgangsstammes könnten einige wenige Zellen sein, deren zufällige Veränderungen im Erbgut zu einer erhöhten Produktbildung führen. Um diese nun zu identifizieren, müssen alle erzeugten Stammvarianten einzeln kultiviert und mithilfe komplizierter, chemisch-analytischer Verfahren in Bezug auf ihre Produktivität untersucht werden – dem sogenannten Screening. Die so isolierten, besten Varianten können in einem weiteren Zyklus von Mutagenese und Screening weiter verbessert werden um so noch leistungsfähigere Mikroorganismen zu erhalten. Prinzipiell kann man diesen sich wiederholenden Zyklus von Mutagenese und Screening so lange durchführen bis man einen für die großtechnische Produktion geeigneten Hochleistungsproduzenten isoliert hat, der sehr effizient einfache und günstige Substrate aus nachwachsenden Rohstoffen zum gewünschten Produkt umsetzen kann (Abb. 1).
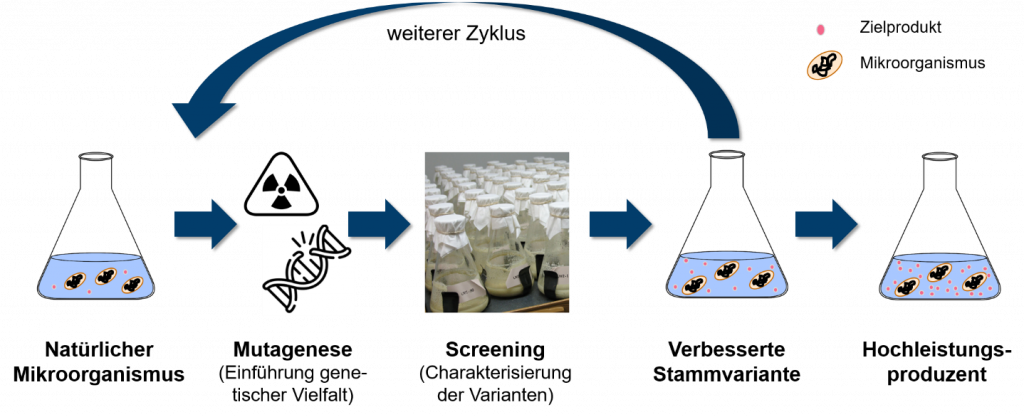
Durch dieses klassische Verfahren zur Stammentwicklung konnten schon viele potente Mikroorganismen isoliert werden, die auch aktuell in der industriellen Produktion eingesetzt werden. Während der Mutagenese-Schritt einfach und kostengünstig durchgeführt werden kann, ist die Charakterisierung jeder einzelnen Stammvariante im Screening-Schritt, mühsam, personalintensiv und sehr zeitaufwendig – kurzum: sehr teuer. Oft müssen viele zehntausend Varianten untersucht werden, bis man einen geeigneten Kandidaten identifiziert hat. Diese gewünschten Varianten könnten wesentlich einfacher erkannt werden, wenn sie z.B. farbig wären oder leuchten würden. Dies ist jedoch bei den wenigsten biotechnologisch interessanten Substanzen der Fall. Um nun den Screening-Schritt zu beschleunigen und somit die Stammentwicklungskosten stark zu senken, mussten Wege gefunden werden, um die erhöhte Produktbildung in ein einfaches, idealerweise optisches Signal zu übersetzen, dass sehr schnell maschinell ausgelesen werden kann. Wenn man dies nun auch noch direkt in einzelnen Zellen durchführen könnte, die typischerweise 1 – 2 millionstel Meter groß sind, könnte man sogar die individuelle Kultivierung jeder Stammvariante umgehen und gute Varianten aus einer einzigen Kultur mit vielen Milliarden Zellen isolieren.
Die Biotechnologen des IBG-1 haben sich dieser Herausforderung gestellt und sogenannte transkriptionelle Biosensoren entwickelt, die in Gegenwart des gewünschten Produktes ein Lichtsignal aussenden, wenn sie mit einem Laser bestrahlt werden (Abb. 2a). Im Gegensatz zu den FRET-Sensoren, die auch im IBG-1 konstruiert werden und die spezielle Bindeproteine nutzen, machen sich die Wissenschaftler bei transkriptionellen Biosensoren zelleigene genetische Schaltkreise zunutze. Diese werden so umprogrammiert, dass sie direkt in den Zellen die Bildung stark fluoreszierender Proteine anschalten, wenn erhöhte Produktkonzentration vorliegen. Diese sogenannten Reporterproteine senden Licht aus, sobald sie mit Laserlicht einer bestimmten Wellenlänge angeregt werden. Die Spezifität von Biosensoren ist dabei abhängig vom gewählten genetischen Schaltkreis. Aufgrund der großen natürlichen und synthetischen Vielfalt dieser Schaltkreise, können aber generell Biosensoren für eine große Vielfalt von interessanten biotechnologischen Produkten konstruiert werden. Interessanterweise liefern viele Biosensoren nicht einfach nur ein AN/AUS Signal, sondern geben eine Fluoreszenzantwort deren Stärke die Produktkonzentration in der Zelle widerspiegelt. Somit kann man auch produzierende Stammvarianten noch einmal genauer unterscheiden und nur mit den besten Kandidaten weiterarbeiten.
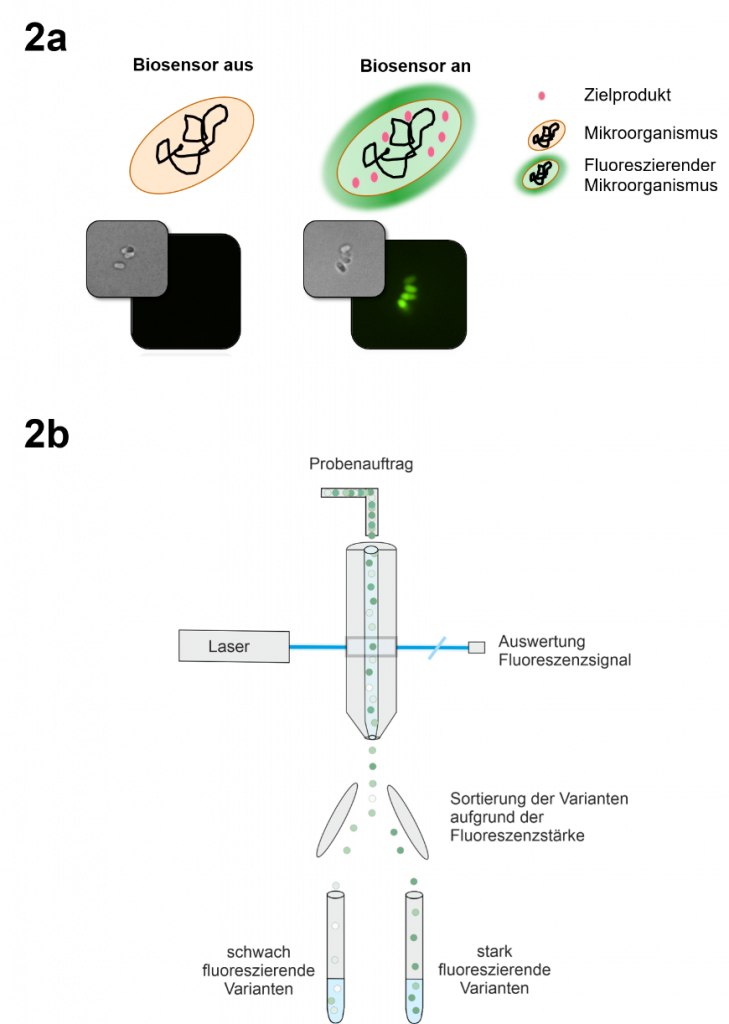
Ohne ein geeignetes Gerät zur Identifizierung und Isolierung einzelner, stark fluoreszierender Zellen ist aber noch nichts gewonnen. In Jülich wird deshalb die Biosensortechnologie mit einem Gerät zur fluoreszenzaktivierten Zellsortierung (FACS, engl. fluorescence activated cell sorting) kombiniert (Abb. 2b). In einem solchen Analysegerät werden einzelne Zellen in einem dünnen Flüssigkeitsstrom an einem Laserstrahl vorbeigeleitet. Ist der Biosensor angeschaltet und das Reporterprotein ist vorhanden, wird von der Bakterienzelle ein Fluoreszenzsignal abgegeben, das ein Detektor im FACS-Gerät erkennen und quantifizieren kann. Anhand der Signalstärke können stark fluoreszierende Zellen also vom Gerät erkannt und direkt gesammelt werden, damit diese dann entweder genauer untersucht oder direkt einem weiteren Mutagenese/Screening Zyklus zugeführt werden können. Ein FACS-Gerät arbeitet außergewöhnlich schnell. Typischerweise können über 15.000 Zellen pro Sekunde bezüglich ihrer Fluoreszenzeigenschaften untersucht werden. Damit können also innerhalb weniger Minuten und ohne großen Aufwand mehr Stammvarianten untersucht werden als in mehreren Monaten ohne diese Technologie.
Somit sind Biosensoren in Kombination mit Zellsortierern ein wertvolles und effizientes Werkzeug für die Stammentwicklung und werden in Zukunft die Entwicklung nachhaltiger Produktionsprozesse entscheidend beschleunigen – Stammentwicklung auf der Überholspur eben!
Ein Beitrag von Christiane Sonntag und Jan Marienhagen.

No Comments
Be the first to start a conversation